Die Analyse von Umweltproben ist entscheidend, um die komplexen Zusammenhänge in natürlichen und künstlichen Ökosystemen zu verstehen. Im Zentrum dieser Forschung steht das Umweltlabor, das sich auf die Charakterisierung einzelner Zellen in mikrobiellen Populationen oder komplexen mikrobiellen Gemeinschaften spezialisiert hat. Durch modernste Technologien wie die Durchflusszytometrie werden nicht nur die Zellheterogenität reiner Populationen erfasst, sondern auch dynamische Veränderungen in der Gemeinschaftszusammensetzung über die Zeit hinweg routinemäßig analysiert. Diese Daten sind unerlässlich, um die Prinzipien der Populations- und Gemeinschaftsökologie zu entschlüsseln und Wachstumsraten indigener Zelltypen zu bestimmen. Ein tiefgreifendes Verständnis dieser Prozesse trägt maßgeblich zum Umweltschutz und zur Entwicklung nachhaltiger Lösungen bei.
Parallel zu den Lichtstreueigenschaften können im Umweltlabor autofluoreszierende Pigmente und Proteine pro Zelle quantifiziert werden. Darüber hinaus ermöglichen mit fluoreszierenden Farbstoffen extrinsisch gefärbte Vitalitätszustände oder andere Zelleigenschaften die Bestimmung verschiedener physiologischer Zustände. Es können Zellen aus nahezu jeder Umgebung analysiert werden, von Wasserproben bis hin zu Bodenmikroorganismen. Zur Auswertung dieser umfangreichen Daten stehen fortschrittliche Bioinformatik-Tools zur Verfügung. Ein weiterer entscheidender Vorteil ist die fluoreszenzaktivierte Zellsortierung (FACS), mit der Zelluntergruppen unterschiedlicher physiologischer Zustände oder spezifische Zelltypen aus Gemeinschaften isoliert werden können. Dies ermöglicht weitere Downstream-Analysen wie Sequenzierung (16S rRNA-Gensequenzierung oder Genomsequenzierung) oder proteomische Analysen. Die Zellen können auch in definierten Mengen (zwischen 1 und 1000) in Wellplatten zur Weiterkultivierung oder auf Mikroskopobjektträger zur Visualisierung gezählt und sortiert werden.
Das Prinzip der Durchflusszytometrie im Umweltlabor
Die Durchflusszytometrie ist eine auf UV- und sichtbarem Licht basierende mikroskopische Technologie, die eine multiparametrische Analyse von Zellen ermöglicht. Dabei passieren die Zellen als einzelne Ereignisse in einem Flüssigkeitsstrom das Objektiv. Die hydrodynamische Fokussierung gewährleistet, dass die Zellen präzise ausgerichtet und als Einzelzellen für eine genaue Analyse bereitgestellt werden. Während die Zellen den Detektionsbereich passieren, werden sie von Photonen getroffen, die von einem oder mehreren Lasern emittiert werden. Die Zellen streuen oder emittieren Photonen, die dann von Photomultiplier-Röhren (PMTs) erfasst werden. Diese erzeugen einen verstärkten elektrischen Strom, der mit den Fluoreszenzintensitäten oder Streueigenschaften der Zellen korreliert ist.
Photonen, die im Winkel von weniger als 20⁰ nach vorne gestreut werden, erzeugen das Vorwärtsstreulicht-Signal (Forward Scatter); dieser Parameter korrespondiert mit der Zellgröße. Orthogonal gestreute Photonen, die in einem Winkel von 90⁰ erfasst werden, erzeugen das Seitwärtsstreulicht-Signal (Side Scatter); dieser Parameter korrespondiert mit der Zellgranularität. Zusätzlich können fluoreszierende Lichtsignale mittels Durchflusszytometrie, ebenfalls in einem Winkel von 90°, erfasst und durch eine Reihe von Filtern geleitet werden, um die Fluoreszenzemission spezifisch zu messen. Dies liefert verschiedene zellgebundene Informationen, die zur Unterscheidung verschiedener Zelltypen in Populationen oder Gemeinschaften einer Probe verwendet werden können. Die Zellsortierung (FACS) wird für die präzise Isolation einzelner Zellen eingesetzt. Bei dieser Methode werden stromabwärts der Detektionszone Tröpfchen gebildet, die die Zielzellen enthalten. Diese Tröpfchen werden dann entweder positiv oder negativ geladen und durch Ablenkplatten voneinander getrennt. Jedes spezifische Merkmal, wie Fluoreszenzintensität oder Zellgröße, kann für eine Sortierungsentscheidung herangezogen werden. Vitale Zellen können auf Agarplatten, in Wells oder Röhrchen mit geeigneten Wachstumsmedien sortiert werden. Es ist auch möglich, Zellen für weitere Downstream-Analysen zu sortieren, z. B. für die Metagenom-Sequenzierung spezifischer Zellsubsets. Die fortschrittliche Technologien für die Umwelt ermöglicht hierbei präzise und zuverlässige Ergebnisse.
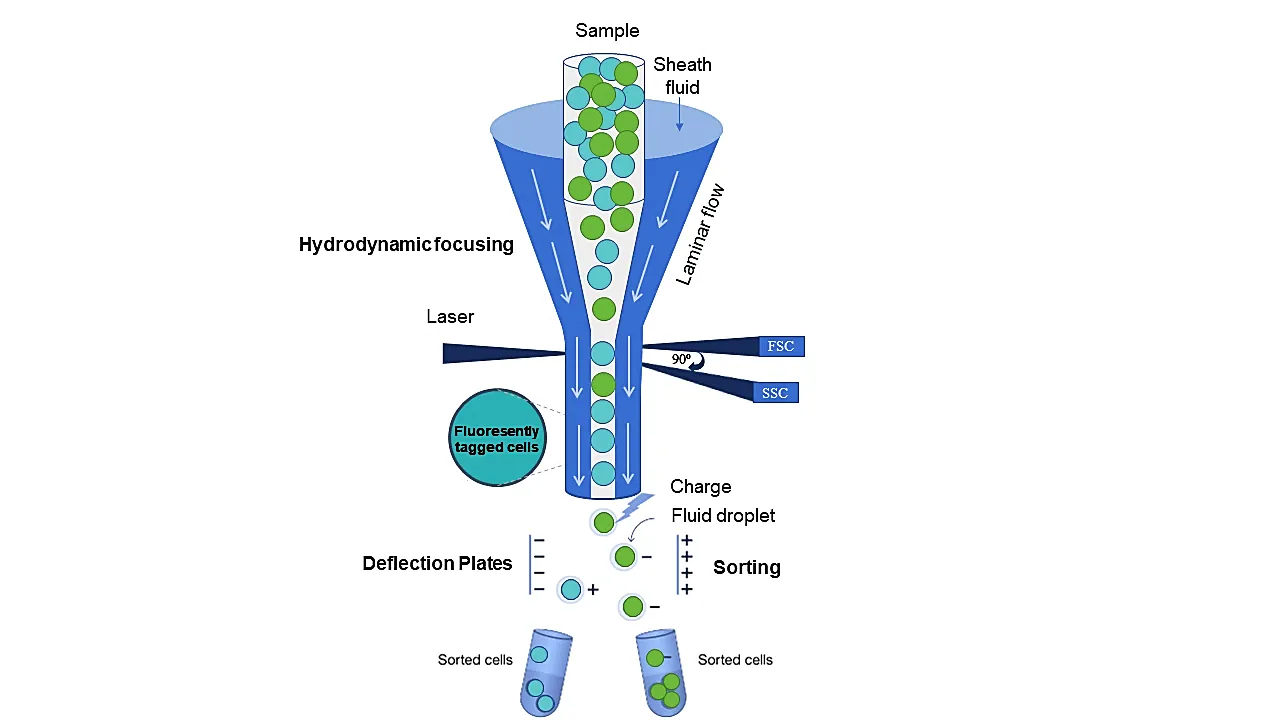 Prinzip der Durchflusszytometrie für Umweltanalysen
Prinzip der Durchflusszytometrie für Umweltanalysen
Modernste Ausstattung für Ihr Umweltlabor
Unser Umweltlabor verfügt über fünf Durchflusszytometer, die für verschiedene Forschungsaktivitäten eingesetzt werden. Drei davon werden zu Sortierungszwecken und zwei als Tischgeräte verwendet. Eine vergleichende Übersicht unserer aktuellen Geräte finden Sie in der folgenden Tabelle.
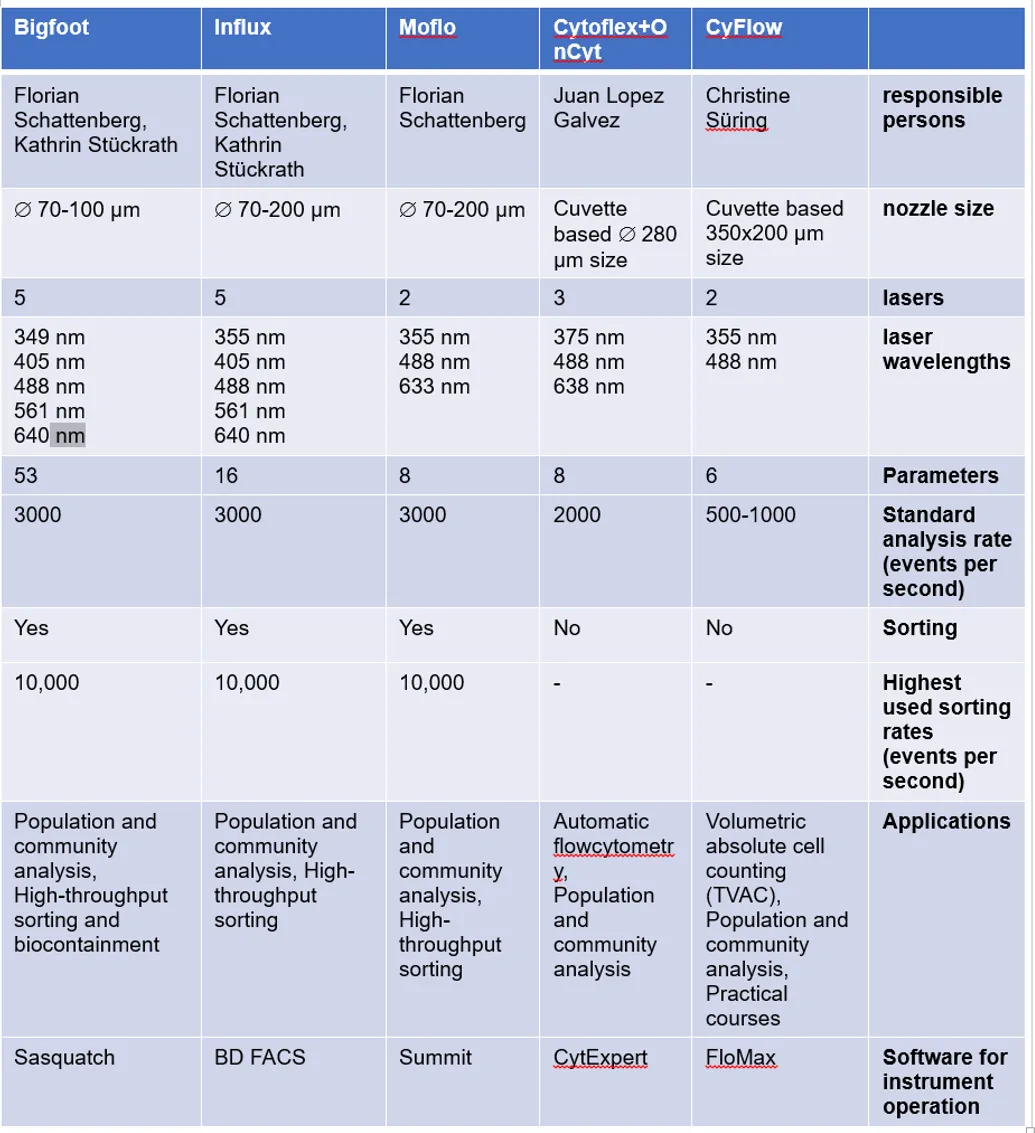 Übersicht der Durchflusszytometer-Ausstattung
Übersicht der Durchflusszytometer-Ausstattung
Bigfoot Spektral-Zellsorter
Mit bis zu 5 Lasern und 53 Detektoren zeichnen sich die spektralen Entmischungsfähigkeiten des Bigfoot-Geräts durch eine hohe Diskriminierung von Zelltypen in Populationen und Gemeinschaften aus. Es kann Autofluoreszenz und entsprechende Veränderungen definierter und empfindlicher als andere Geräte detektieren. Das Biosicherheitslevel-2-Containment mit integriertem HEPA-Filter und Aerosolmanagement bietet integrierte Biokontaminationseindämmung und Personenschutz. Dies macht Hochleistungs- und Hochdurchsatzmessungen für Downstream-OMICS-Anwendungen bequemer und schneller. Der Bigfoot Spektral-Zellsorter ermöglicht Multiway-Sortierungen in 1,5 mL bis 50 mL Röhrchen, in 24 bis 384-Well-Platten und sogar auf 1536-Well-Sortierplatten.
BD Influx™ Zellsorter
Der BD Influx™ Zellsorter verfügt über 5 Anregungslaser, die mehrere Zellparameter gleichzeitig erfassen können. Die Detektion geringer Fluoreszenzintensitäten ermöglicht eine besonders gute Charakterisierung von Bakterien und eine hochauflösende Analyse mikrobieller Populationen und Gemeinschaften. Er nutzt die BD FACS™ Accudrop-Technologie zur präzisen Tropfenverzögerungsbestimmung für die Sortierung direkt auf Kulturplatten, 24- bis 96-Well-Platten und 1,5 – 50 mL Röhrchen. Mehrere 4-Wege- und 6-Wege-Sortierungen sind möglich und essentiell für umfassende Umweltanalysen.
MoFlo Legacy Zellsorter
Der MoFlo Legacy Zellsorter ist mit drei Lasern ausgestattet und wird sowohl für Subpopulations- als auch für Gemeinschaftsanalysen eingesetzt. Es handelt sich um ein älteres Gerät, das demnächst abgebaut wird, aber über Jahre hinweg wichtige Beiträge zur Umweltforschung geleistet hat.
CyFlow Space
Im Vergleich zu anderen Zytometern ist der CyFlow Space relativ einfach zu bedienen und bietet eine Auflösung von 0,5 bis 100 µm für die Partikelmessung. Er analysiert die absolute Zell- und Partikelanzahl pro mL mittels True Volumetric Absolute Counting (TVAC). Dieses Gerät wird routinemäßig für die Analyse von Umweltproben wie Abwasser und zur Ausbildung von Studierenden in Praktika und Hochschulpraktika eingesetzt. Dies unterstreicht seine Bedeutung als unverzichtbares Werkzeug im Umweltlabor.
CytoFLEX
Der CytoFLEX (Beckman Coulter, Brea, CA, USA) enthält 3 Laser und ist mit einem OC-300 Automatisierungsgerät (onCyt Microbiology, Zürich, Schweiz) für automatisierte Durchflusszytometrie-Operationen wie Probenahme, Fixierung und Färbung gekoppelt. Das Gerät ist eine Leihgabe von OnCyte und wird nur bis Mitte 2025 zur Verfügung stehen. Es misst die Zelldichte oder Fingerprint-ähnliche Muster bakterieller Populationen oder Gemeinschaften und generiert somit geeignete Daten für leistungsstarke automatische Datenanalyse- und Interpretationspipelines. Dies macht die Daten in kürzester Zeit verfügbar und ermöglicht eine Online-Überwachung von Bioreaktoren, was insbesondere im Umweltmonitoring von großer Relevanz ist.
Methoden im Umweltlabor: Probenvorbereitung und Färbung
Die Durchflusszytometrie-Gruppe ist spezialisiert auf die Analyse von prokaryotischen (Bakterien, Cyanobakterien und Archaeen) und eukaryotischen (Hefen und Algen) Zellen. Mikroorganismen können in allen Lebensstadien analysiert werden, wie vegetativen/toten Zellen, Sporen, Biofilmen etc. Für die Analyse sind Suspensionen mit Einzelzellen erforderlich. Bei allen Proben wird zusätzlich eine Filtration mit Celltrics-Filtern mit einer Porengröße von 50 µm durchgeführt, um ein Verstopfen der 70 µm Düse zu verhindern.
Lebende versus fixierte Zellen
Die Durchflusszytometrie kann sowohl mit lebenden (Echtzeit-) als auch mit fixierten Zellen durchgeführt werden. Die Fixierung mit Chemikalien wie Paraformaldehyd (PFA) vernetzt intensiv Oberflächenproteine von Zellen, was ihre Membranen stabilisiert und verschließt. Nach 30 Minuten wird das PFA entfernt und die Zellen in 70 % Ethanol suspendiert und bei -20°C gelagert. Diese Methode ermöglicht die Lagerung von Proben über lange Zeiträume von bis zu 6 Monaten. Zudem bestehen bei fixierten Proben keine Biosicherheitsbedenken. Ähnliche Färbeprotokolle können sowohl für lebende als auch für fixierte Zellen verwendet werden, um spezifische zelluläre Komponenten oder Biomarker zu markieren. Lebende Zellen sind metabolisch aktiv und behalten ihre physiologischen Funktionen bei, während fixierte Zellen ihre Stoffwechselaktivität eingestellt und die Zellstruktur konserviert haben. Zur Fixierung von Zellen stehen zwei Protokolle zur Verfügung.
 Protokolle zur Zellfixierung
Protokolle zur Zellfixierung
Fluoreszierende Färbemittel und Proteine
Die Analyse im Umweltlabor nutzt eine Vielzahl von Fluoreszenzmarkern, um zelluläre Eigenschaften sichtbar zu machen.
1. Fluoreszierende Proteine
Fluoreszenzmarkierte Reporterproteine können leicht gemessen werden. Einige der weit verbreiteten fluoreszierenden Proteine sind:
- Grün fluoreszierendes Protein (GFP): GFP ist eines der häufigsten fluoreszierenden Proteine, das als Marker für die Genexpression in der Molekular- und Zellbiologie verwendet wird. Die Anregungs- und Emissionswellenlängen für GFP liegen bei 470 bis 490 nm bzw. 510 bis 530 nm.
- Rot fluoreszierendes Protein (RFP): RFPs, wie DsRed und mCherry, emittieren rotes Licht und werden oft zusammen mit GFP für Dual-Labeling-Experimente verwendet. RFPs haben typischerweise eine Anregungswellenlänge im Bereich von 550 bis 590 nm und eine Emissionswellenlänge um 590 bis 650 nm.
- Gelb fluoreszierendes Protein (YFP): YFP, wie Citrine (abgeleitet von Aequorea victoria), emittiert gelbes Licht und wird häufig in Kombination mit anderen fluoreszierenden Proteinen eingesetzt. YFP hat eine Anregungswellenlänge, die typischerweise von 505 bis 515 nm reicht, und eine Emissionswellenlänge, die typischerweise von 520 bis 570 nm reicht.
- Cyan fluoreszierendes Protein (CFP): CFPs, wie Cerulean (abgeleitet von Aequorea victoria), emittieren cyanfarbenes Licht und werden in Multicolor-Imaging-Experimenten eingesetzt. CFP hat eine Anregungswellenlänge von etwa 433 nm und eine Emissionswellenlänge von etwa 475 nm.
- Blau fluoreszierendes Protein (BFP): BFPs emittieren blaues Licht und können in Kombination mit anderen fluoreszierenden Proteinen für Multicolor-Imaging verwendet werden. BFPs haben eine Anregungswellenlänge im Bereich von etwa 380 bis 410 nm und eine Emissionswellenlänge um 440 bis 470 nm.
2. Autofluoreszenz
Einige Bakterien, Cyanobakterien, Algen und Archaeen enthalten endogene Fluorophore, die bei Anregung durch UV/sichtbare Strahlung einer geeigneten Wellenlänge Autofluoreszenz emittieren. Verschiedene zelluläre Komponenten können zur Autofluoreszenz beitragen, einschließlich Pigmenten. Die Autofluoreszenz-basierte Identifizierung ermöglicht eine markierungsfreie Detektion, ohne die Notwendigkeit exogener Fluoreszenzmarker oder -sonden. Dies ist besonders wertvoll, um ein Bild Umweltschutz auf molekularer Ebene zu erhalten.
3. Fluoreszenzfarbstoffe
Zellen können durch eine Vielzahl von fluoreszierenden Reagenzien für die Durchflusszytometrie-Analyse gefärbt werden. Dazu gehören DNA-bindende Farbstoffe, Vitalitätsfarbstoffe und Ionenindikatorfarbstoffe. In der Durchflusszytometrie-Gruppe werden die folgenden Fluoreszenzfarbstoffe für die Populations- und Gemeinschaftsanalyse verwendet:
- DAPI (4′,6-Diamidino-2-phenylindol): DAPI ist ein DNA-spezifischer Fluoreszenzfarbstoff, der zur Färbung bakterieller DNA verwendet werden kann. Er wird häufig zur Beurteilung des Zellzyklusfortschritts und des Community-Fingerprintings eingesetzt.
- SYTO 9: SYTO-Farbstoffe sind Nukleinsäurefarbstoffe, die intakte bakterielle Zellen penetrieren können. Sie werden oft zur Gesamtzellzählung und Vitalitätsbewertung verwendet. SYTO 9 ist ein grün fluoreszierender Farbstoff, der sowohl lebende als auch tote Zellen färbt.
- Propidiumiodid (PI): PI ist ein DNA-bindender Farbstoff, der zur Differenzierung zwischen lebenden und möglicherweise toten Zellen basierend auf ihrer Membranintegrität verwendet werden kann. Lebende Zellen schließen PI aus, während Zellen mit beeinträchtigten Membranen PI eindringen lassen.
- SYBR Green: SYBR Green ist ein Nukleinsäure-bindender Farbstoff, der bei Bindung an doppelsträngige Nukleinsäuren Fluoreszenz emittiert. SYBR Green bindet sowohl an DNA als auch an RNA.
- Hoechst 33342: Hoechst 33342 ist ein zellpermeabler DNA-Farbstoff, der bevorzugt an Adenin-Thymin (AT)-reiche Regionen der DNA bindet. Er wird zur Quantifizierung des DNA-Gehalts und zur Identifizierung verschiedener Phasen des Zellzyklus verwendet.
- FDA (Fluoresceindiacetat) ist ein nicht-fluoreszierender, zellpermeabler Farbstoff, der über intrazelluläre Esterasen in lebenden Zellen in ein grün fluoreszierendes Produkt, Fluorescein, umgewandelt wird. Tote Zellen zeigen keine Esterase-Aktivität, daher wird der Farbstoff zur Beurteilung der Zellvitalität verwendet.
- 3,3′-Dihexyloxacarbocyaniniodid (diOC6(3)) ist ein lipophiler kationischer Farbstoff, der Veränderungen des bakteriellen Membranpotenzials anzeigt und Einblicke in die bakterielle Vitalität, Membranpotenzialaktivität und Reaktionen auf verschiedene Umweltbedingungen geben kann.
- Nilrot ist ein lipophiler Farbstoff, der широко zur Färbung und Visualisierung neutraler Lipide, insbesondere Triglyceride und Lipoproteine, in Zellen verwendet wird.
- Lektine oder Antikörper, die mit fluoreszierenden Farbstoffen konjugiert sind, können zur Identifizierung spezifischer Zellpopulationen basierend auf Kohlenhydrat-vermittelten Interaktionen oder zur Bindung an spezifische Zelloberflächenregionen verwendet werden.
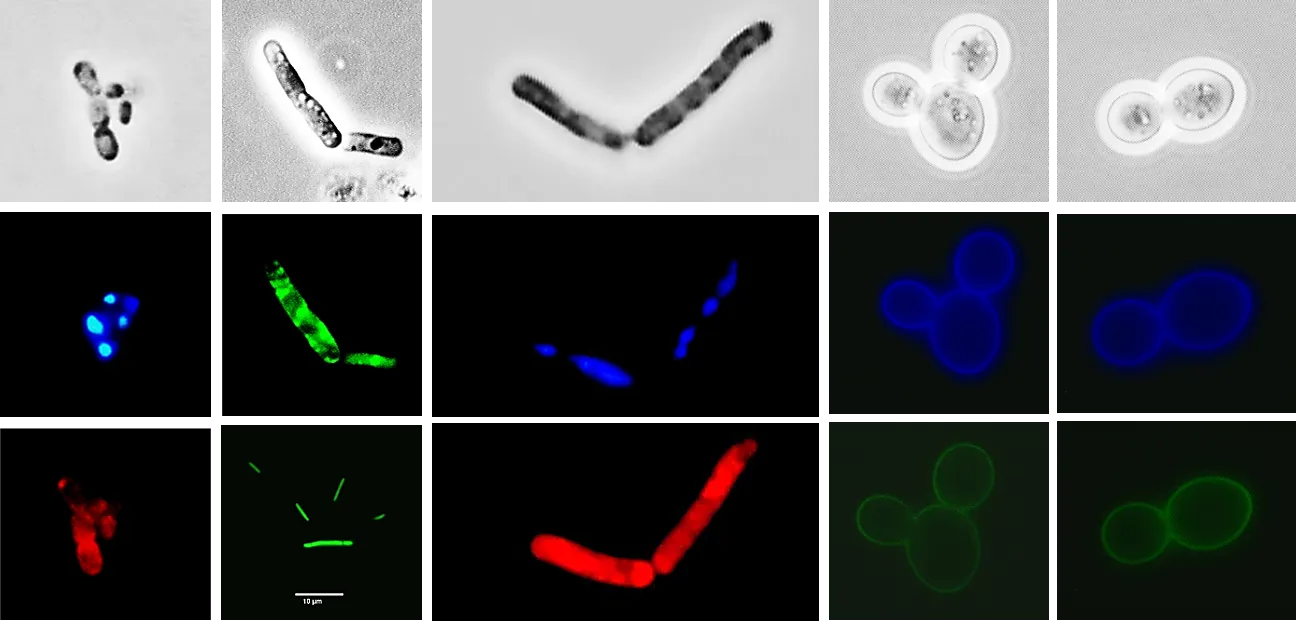 Mikroskopische Ansicht gefärbter Zellen
Mikroskopische Ansicht gefärbter Zellen
Ausrichtung der Durchflusszytometer und Mock Communities
Im Umweltlabor können Rauschen durch unpräzise Ausrichtung der Optik und Schwankungen der Laserstabilität über die Zeit entstehen. Um dies zu vermeiden, werden Laser sorgfältig ausgerichtet, um sicherzustellen, dass ihre Strahlen für die Detektion der Fluoreszenzemission korrekt auf den Probenstrom fokussiert sind. Standardisierte monodisperse Beads werden routinemäßig in den Durchflusszytometern eingesetzt. Typischerweise werden mono- oder multifluoreszierende Beads mit einer Größe von 0,5 bis 1 μm für diesen Zweck verwendet. Diese Kalibrierungsbeads sind fluoreszierende Mikrosphären mit bekannten Fluoreszenzeigenschaften, einschließlich Fluoreszenzintensität und Emissionsspektren. Durch das Laufen von Kalibrierungsbeads durch das Durchflusszytometer können Operatoren PMT-Spannungen und Verstärkungseinstellungen anpassen, um Signal-Rausch-Verhältnisse zu standardisieren und zu optimieren. Eine ordnungsgemäße Ausrichtung mittels Kalibrierungsbeads verbessert die Zuverlässigkeit und Reproduzierbarkeit von Durchflusszytometrie-Daten und ermöglicht eine genaue Quantifizierung und Analyse von Proben. Für Präsentationen zum Thema Umwelt sind solche präzisen Methoden oft ein zentraler Bestandteil.
Während Alignment-Beads dazu beitragen, instrumentelles Rauschen zu minimieren und die allgemeinen Maschineneinstellungen zu harmonisieren, ist die Verwendung einer künstlichen mikrobiellen zytometrischen Mock-Community (mCMC) unerlässlich für die Validierung von Labor-Workflows, Färbeprotokollen und den Datenvergleich über Experimente hinweg. Die mCMC fungiert als wichtiger Referenzstandard für die konsistente Charakterisierung mikrobieller Gemeinschaften mittels zytometrischer Analyse, insbesondere in Längsschnittstudien. Die mCMC, bestehend aus drei reinen Stämmen (Stenotrophomonas rhizophila DSM 14405, Kocuria rhizophila DSM 348 und Paenibacillus polymyxa DSM 36), zeigt Zellpopulationen, die über das 2D-Plot verteilt sind und sowohl den unteren als auch den oberen logarithmischen Skalenbereich abdecken, wodurch ihre Trennung durch Gates ermöglicht wird. Darüber hinaus berücksichtigt die mCMC die Heterogenität, die sich aus Subpopulationen ergibt, was die Analyse komplexer macht. Ihre Nützlichkeit erstreckt sich auf die Anpassung der Bakterienposition und des Auflösungsbereichs in allen kommerziellen Durchflusszytometern, die Verbesserung der Instrumentenauflösung und die Reduzierung von Hintergrundrauschen, die Identifizierung und Minderung bedienerabhängiger Fehler oder Variationen in der Zellbehandlung sowie die Aufrechterhaltung konsistenter Zellpositionen während Langzeitexperimenten, um die Stabilität zytometrischer Gemeinschaftsmuster zu gewährleisten und somit automatisierte bioinformatische Auswertungs-Pipelines zu erleichtern.
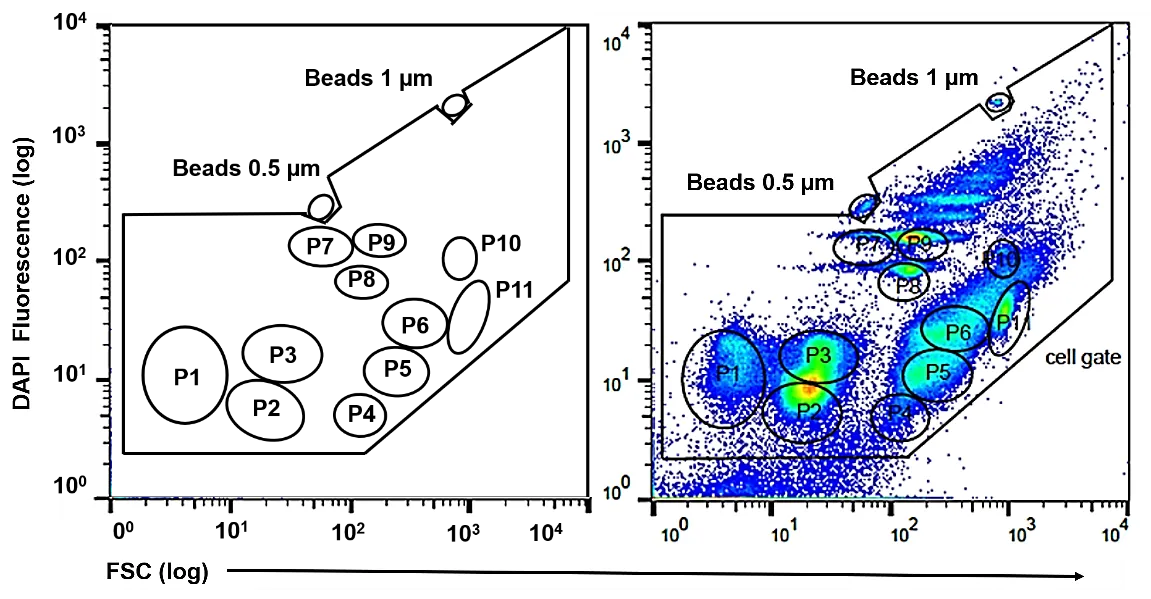 Zell-Gate und Gate-Vorlage der mCMC-Platte
Zell-Gate und Gate-Vorlage der mCMC-Platte
Bioinformatische Analysetools für Umweltproben
Um künstliche oder natürliche Gemeinschaften in gemanagten Systemen zu untersuchen, zu überwachen und letztendlich zu kontrollieren, sind schnelle Detektions- und Bewertungsmethoden erforderlich. Zytometrische Fingerabdrücke komplexer mikrobieller Gemeinschaften und reiner Kultur-Subpopulationen können mit dem grafischen Benutzerinterface (GUI) des biTCa Analyze Tools ausgewertet werden. Es enthält alle von unserem Umweltlabor entwickelten bioinformatischen Tools wie Cytometric Histogram Image Comparison (flowCHIC), Cytometric Barcoding (flowCyBar) und flowEMMi.
1. Cytometric Histogram Image Comparison (flowCHIC)
Alle zytometrischen 2D-Plots werden in Graustufenbilder umgewandelt, und diese Bilder werden dann basierend auf dem durchschnittlichen Grauwert pro informativem Pixel verglichen. Die Unterschiede in der Zellhäufigkeit werden durch NMDS-Plots dargestellt. Das R-Paket flowCHIC und seine Bedienungsanleitung können von der Bioconductor-Plattform heruntergeladen werden.
2. Cytometric Barcoding (flowCyBar)
flowCyBar veranschaulicht die Veränderungen in den Zellhäufigkeiten von Subgemeinschaften in Korrelation zu zeitlichen, räumlichen, abiotischen und biotischen Parametern mittels NMDS, Boxplots und Heatmaps (Korrelation, Interaktionspotential). Das R-Paket flowCyBar und seine Bedienungsanleitung können von der Bioconductor-Plattform heruntergeladen werden.
3. flowEMMi
flowEMMi v2 ist ein automatisches modellbasiertes Clustering-Tool für zytometrische Gemeinschaftsdaten, das auf multivariaten Gaußschen Mischmodellen basiert. Es markiert alle Subgemeinschaften in den Histogrammen mit verschiedenen Gates, und dann wird eine kombinierte Gate-Vorlage schließlich auf alle Proben angewendet. Dieses Tool bietet schnelles und genaues Zell-Clustering für dynamische Gemeinschaftsanalysen, wobei technisches Rauschen oder Beads umgangen werden.
Anwendungen im Umweltlabor
Die fortschrittlichen Methoden der Durchflusszytometrie finden vielfältige Anwendungen in der Umweltforschung und -überwachung, was unser Umweltlabor zu einem zentralen Akteur macht.
1. Ökologie mikrobieller Gemeinschaften
Die Aufrechterhaltung der gewünschten mikrobiellen Gemeinschaft in Bioreaktoren und die Bestimmung der Strukturen von Gemeinschaften in natürlichen Biosphären sind wichtige Anwendungen der mikrobiellen Durchflusszytometrie. Mikrobielle Gemeinschaften können sehr vielfältig sein, anfällig für stochastische Schwankungen, und der Verlust von Schlüsselakteuren ist häufig. Unsere durchflusszytometrische Analyse in Kombination mit Bioinformatik- und ökologischen Tools enthüllt die Dynamik und das Schicksal von Gemeinschaftsstrukturen viel schneller als Sequenzierungstechnologien. Wir verwenden verschiedene Diversitätsindizes und Interaktionsprofile, um Gemeinschaftsbildungsstrategien und die funktionellen Abhängigkeiten der Gemeinschaftsmitglieder zu beschreiben. Wir bieten diese Expertise an, um das ökologische Paradigma zu verstehen, das die interessierenden Gemeinschaften in ihren jeweiligen Umgebungen formt. Hierbei spielen Visualisierungen im Umweltschutz eine große Rolle.
Zu beantwortende Fragen:
- Analyse der Variation der intrakommunalen Struktur von Mikroorganismen und Identifizierung der Subgemeinschaftsfunktion.
- Ermöglichung der Quantifizierung von Einzelzelldynamiken und der Online-Bewertung von Gemeinschaftsmerkmalen.
- Berechnung der Stabilitätseigenschaften (Resistenz, Resilienz, Verdrängungsgeschwindigkeit und Elastizität) der Bioreaktor-Gemeinschaft.
- Interpretation der Gemeinschaftsparadigmen, Trends und Entwicklungen.
- Aufdeckung möglicher Stimuli und Einschränkungen, die die Gemeinschaftsstruktur und -funktion beeinflussen.
- Aufrechterhaltung einer stabilen Gemeinschaft in Ihrem Bioreaktor, Vermeidung von Auswaschung.
Vorgehensweise:
Zellen müssen fixiert werden. Die Zelldichte muss angepasst, eine DAPI-Färbung durchgeführt und ein Fingerprinting vorgenommen werden. Die resultierenden Subgemeinschaften müssen gegatet und die Häufigkeit der Zellen pro Gate bestimmt werden. Jedes Bewertungstool kann dann verwendet werden.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension ohne Verunreinigungen sein. Die Zellen können sonifiziert, gevortexed, gewaschen und filtriert werden, um Aggregate und Partikel zu entfernen.
Entwickelte Technologien:
- Geschlossene Massentransfertechnologie: Unsere kürzlich entwickelte geschlossene Massentransfertechnologie stellt sicher, dass die mikrobiellen Gemeinschaften stabil gehalten und in den Bioreaktoren verbleiben. Wenn Sie mit dem Verlust mikrobieller Gemeinschaften in Ihrem Bioprozess zu kämpfen haben, könnte dies eine alternative Lösung sein. Kontaktieren Sie uns.
- Stabilitäts- und Diversitätsmaße: Ökologische Stabilitätseigenschaften mikrobieller Gemeinschaften können mittels Durchflusszytometrie bewertet werden.
- Netto-Wachstumsrate: Die Bestimmung des Wachstums von Subpopulationen innerhalb von Gemeinschaften kann mittels Durchflusszytometrie bewertet werden.
2. Differentialfärbung und Subpopulationsanalyse
Die Biokonversion kann durch die Analyse verschiedener Subpopulationen und ihrer Beiträge verbessert werden. Dies ermöglicht die Prozessoptimierung durch Änderung operativer Parameter. Für die differenzierte Färbung von Zellen aus reinen Kulturen oder Kokulturen steht eine Reihe von Fluoreszenzfarbstoffen zur Verfügung für:
- Biomolekülsynthese
- Metabolisch aktive Zustände
- Zellzyklusstudien
- Subpopulationsanalyse
Zu beantwortende Fragen:
- Studium verschiedener physiologischer oder expressioneller Subpopulationen des interessierenden Organismus.
- Zellzyklusmerkmale des Organismus in Abhängigkeit von verschiedenen Substraten oder Einschränkungen.
- Vitalität und Zählung von lebenden/toten Zellen.
- Polymerbildung wie Polyhydroxyalkanoate (PHA).
- Fluoreszenzmarkierte Genexpression in Subpopulationen.
Vorgehensweise:
Zellen können lebend oder fixiert gemessen werden (mit den etablierten Fixierungsprotokollen). Nach optischer Dichteanpassung, Färbung und Filtration werden die Zellen zytometrisch analysiert.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension ohne Verunreinigungen sein, die sonifiziert, gevortexed, gewaschen und filtriert werden kann, um Aggregate und Partikel zu entfernen. Die gewonnenen Erkenntnisse sind ein wichtiger Beitrag zum Thema Umweltschutz und Nachhaltigkeit.
3. Sortierung für OMICS-Anwendungen
Die Arbeitsgruppe Durchflusszytometrie verfügt über drei Sorter. Einzelzellen können anhand ihrer Fluoreszenzmerkmale voneinander differenziert und sortiert werden. Ausgewählte Zellen werden für eine Reihe von Downstream-Experimenten verwendet, darunter funktionelle Assays, DNA-Analyse, Gen-/Proteinexpressionsanalyse, Reinkulturen, Koloniecharakterisierung und Stammentwicklung.
Zu beantwortende Fragen:
- Trennung von fluoreszenzemittierenden Reporterzellen, die das Protein von Interesse exprimieren, von Hintergrundzellen.
- Sortierung von differenziell gefärbten Zellen voneinander und von nicht gefärbten Zellen.
- Sortierung verschiedener physiologischer Zustände von Zellen während der Replikation zur Rekultivierung.
- Trennung und Sortierung von gering abundanten Zellen von hoch abundanten Zellen für die 16S rRNA-Sequenzierung oder die Genomsequenzierung oder andere OMICS-Anwendungen.
- Sortierung verschiedener Zellen und Sporen basierend auf Größe und Dichte.
- Sortierung von Archaeen/Cyanobakterien von Bakterien basierend auf ihrer Autofluoreszenz.
- Sortierung von lebenden Zellen von toten Zellen.
Vorgehensweise:
Identifizierung des interessierenden Sortierbereichs durch Gating. Bitte kontaktieren Sie die verantwortliche Person für einige anfängliche Testläufe.
Anforderungen an die Probe:
Die Probe muss eine Zellsuspension mit Zelldichten von nicht weniger als 10⁵ sein. Die Zellen können sonifiziert, gevortexed, gewaschen und filtriert werden, um Aggregate und Verunreinigungen zu entfernen. Das Volumen hängt von den Downstream-Anwendungen ab.
Fazit: Das Umweltlabor als Innovationsmotor
Das Umweltlabor ist heute mehr denn je ein unverzichtbarer Akteur für die Forschung und Anwendung im Bereich der mikrobiellen Ökologie und Umweltanalytik. Durch den Einsatz modernster Durchflusszytometrie und bioinformatischer Tools können wir tiefgreifende Einblicke in die komplexen Strukturen und Funktionen mikrobieller Gemeinschaften gewinnen, die für unser Ökosystem von entscheidender Bedeutung sind. Von der präzisen Charakterisierung einzelner Zellen bis hin zur detaillierten Analyse ganzer Populationen – die hier vorgestellten Methoden und Geräte ermöglichen es Wissenschaftlern und Forschern, die Dynamik des Lebens auf Mikroebene zu verstehen und wichtige Fragen des Umweltschutzes zu beantworten. Wenn Sie spezifische Fragen zur Analyse von Umweltproben haben oder Unterstützung bei Ihren Forschungsprojekten benötigen, kontaktieren Sie uns gerne. Unser Expertenteam im Umweltlabor steht Ihnen mit Rat und Tat zur Seite, um innovative Lösungen für eine nachhaltige Zukunft zu entwickeln.
